Auf der Suche nach endogenen Proteinen
Dr. Johannes Broichhagen
www.broichhagenlab.com
In seinen Vorlesungen, zusammengefasst in seinem Buch „Was ist Leben?“ beschrieb der Physiker Erwin Schrödinger in den 1940er Jahren, dass „der Ablauf der Lebensvorgänge in einem Organismus […] eine bewundernswerte Regelmäßigkeit und Ordnung [zeigt], die in der unbelebten Materie nicht ihresgleichen findet“.[1]
Knapp 80 Jahre später sind wir heute noch immer auf der Suche nach der Entstehung sowie der Beschreibung von „Leben“, und mit Hilfe von molekularbiologischen (z.B. gene-editing mittels CRISPR/Cas9), massenspektrometrischen (z.B. proteomics, metabolomics), high-throughput sequenzierenden (z.B. single cell RNA sequencing) und hochauflösenden mikroskopischen (z.B. super-resolution STED und STORM) Techniken versuchen wir, die vielen gefundenen Puzzlestücke zu einem regelmäßigen und geordneten Gesamtbild zusammenzusetzen.
Dabei treibt WissenschaftlerInnen nicht nur die fundamentalste Frage nach dem Leben an, sondern im Besonderen die Zusammenhänge und Ursachen in Gesundheits- und Krankheitsbildern, um unter anderem bessere und sichere Therapien und Medikamente zu finden. Durch die Nutzung zahlreicher biologischer Modellsysteme wurde ein großer Wissensfundus geschaffen, doch maßgebliche Fragen sind immer noch schwer zu beantworten, etwa:
- „Wie viele Proteine eines Typs gibt es pro Zelle?“ und
- „Wie sind sie zu einem gegebenen Zeitpunkt und in Abhängigkeit der Zellaktivität räumlich verteilt?“
Dies sind interessante Fragen, vor allem wenn es sich nicht um Modellsysteme handelt, wie etwa Biopsien von Patienten, die nicht (oder sehr schwer) genetisch manipulierbar sind. Um einen tieferen Einblick in die Welt der Biomoleküle, deren Interaktionen, Lokalisationen und Komplexität, zu erhalten, beschäftigt sich unsere Forschungsgruppe mit dem Design und der Benutzung von maßgeschneiderten Sonden (sog. probes) an der Schnittstelle von synthetischer Chemie und angewandter Biologie, im Forschungsfeld der Chemischen Biologie (Chemical Biology).
Richard Dawkins fasste eine wissenschaftliche Aufgabe im Jahr 2013 mit den Worten „Komplexität ist ein Problem, das jede Theorie der Biologie lösen muss“ (engl. „Complexity is a problem that any theory of biology has to solve”[2]) hervorragend zusammen. Auf zellulärer Ebene ist das ein komplexes Orchester an hauptsächlich Proteinen, Zuckern, Fetten und Nukleinsäuren, die sich untereinander durch ihre Wechselwirkungen steuern. Peter Kruse benennt verschiedene (bessere und schlechtere) Lösungsansätze, wie man auf wachsende Komplexität reagiert, und beschreibt eindrücklich: „Ein kompliziertes System kann man vereinfachen durch Trivialisierung, indem man es unterteilt. Ein komplexes System zerstören Sie, wenn Sie trivialisieren.“[3] Was bedeutet dies nun genau für unsere Forschung?
Wir wollen so nah wie möglich an einem komplexen biologischen System wie der Zelle arbeiten, und haben dafür ein paar wichtige Kriterien formuliert: (i) die Untersuchung und Manipulation von Biomolekülen in lebenden (idealerweise Primär-) Zellen (ii) ohne die Verwendung gentechnischer Methoden, die zu Perturbationen führen können (iii) mittels nicht-invasiver Methoden. Dabei bestimmt die Auswahl der Methode die Herangehensweise mit einer minimalen Trivialisierung.
Im Gegensatz zu anderen bildgebenden Verfahren wie etwa Ultraschall oder Kernspintomographie hat die Fluoreszenzmikroskopie durch die Möglichkeit Abbes Gesetz zu umgehen das super-resolution Zeitalter eingeläutet und seither eine Revolution erlebt. Mittels stimulated-emission depletion (STED) oder stochastischer reconstruction (STORM) Mikroskopie können somit zelluläre Auflösungen bis zu 20 Nanometer erreicht werden (zum Vergleich: ein Zellkern besitzt einen Durchmesser von 5–15 Mikrometer, ein globuläres Protein ~5 Nanometer). Beide Systeme benötigen die Einführung von Fluoreszenzfarbstoffen, und es ist kaum ein Zufall, dass die Entwicklung dieser 2014 mit dem Nobelpreis in Chemie honoriert wurde.
Um Proteine spezifisch zu markieren, müssen Fluorophore an ein molekulares Gerüst gekoppelt sein, das sein Ziel exakt erkennt. Traditionellerweise benutzt man hierfür Antikörper-Fluorophor-Konjugate. Diese sind jedoch selbst mit einer gewissen Größe nicht so geeignet, und funktionieren hauptsächlich in fixierten Zellen oder Gewebe, also nicht mehr in lebenden Systemen. Wir versuchen mit moderner synthetischer Chemie, ausgehend von validierter Pharmakologie von Wirkstoffen und mit Hilfe von Kristallstrukturen, individuell designte probes herzustellen. Diese probes finden ihre Zielmolekül spezifisch und können durch das an sie verlinkte Fluorophor per Mikroskopie visualisiert werden.
Das alles funktioniert ohne genetische Manipulation, und bleibt somit so nah wie möglich am nativen System der Zelle. Weiterhin ist die Entwicklung von Farbstoffen ein zentrales Forschungsthema, was Hand in Hand geht mit der Sondenentwicklung. Um dazu je ein Beispiel zu nennen: das Einführen von Deuterium, einem Wasserstoffisotop, in Rhodamin-basierte Farbstoffe zeigt eine Verbesserung der photophysikalischen und chemischen Eigenschaften; die Chromophore sind heller und neigen weniger zu bleichen.[4,5] Dies funktioniert auch wunderbar in der hochauflösenden STED-Mikroskopie, die durch ihre angewandten hohe Laserintensitäten einigen Anspruch an Fluorophore stellt.
Als zweites Beispiel sollen kurz die LUXendine vorgestellt werden.[6,7] Hierbei handelt es sich um spezifische, antagonistische Peptide, die an den glucagon-like peptide-1 Rezeptor (GLP1R) binden, ohne dabei eine physiologische Antwort auszulösen (Abbildung 1A). Damit sind sie hervorragend als probes geeignet. Mittels bioorthogonaler Linker-Chemie konnten diese Peptide mit Farbstoffen, in einer Palette von grün bis zum Nahinfrarotbereich, ausgestattet werden. Dies ermöglicht die Identifizierung dieses wichtigen Rezeptors, der pharmakologisch in der Behandlung von Diabetes angesteuert wird, und lässt eine Vielzahl spannender Experimente zu. Neben super-resolution Mikroskopie, die die Nanodomänen dieses Rezeptors auflöste, konnte man GLP1R in verschiedenen Geweben finden, wie etwa in der Bauchspeicheldrüse und im Gehirn, sowie aufregende Einzelmolekülbildgebung durchführen, in der ein Rezeptor bei seiner Bewegung an der Zelloberfläche nachverfolgt werden kann (Abbildung 1B).
Mit der weiteren Entwicklung solcher Proben, wollen wir in Zukunft das feine Orchester der Zellkommunikation aufklären, und haben hierfür eine Basis an der Schnittstelle der organischen Chemie und Zellbiologie gelegt. Um den Kreis mit Erwin Schrödinger zu schließen, ist die Metapher seiner Katze (Schrödinger’s cat) sehr schön geeignet: sinngemäß weiß man erst, ob die Katze lebt oder tot ist, wenn man den Deckel der Kiste aufmacht, in der sie sitzt. Wir wissen also über manche Zustände erst etwas, wenn man nachschauen kann – und um nachschauen zu können, entwickeln wir neue und smarte Tools.
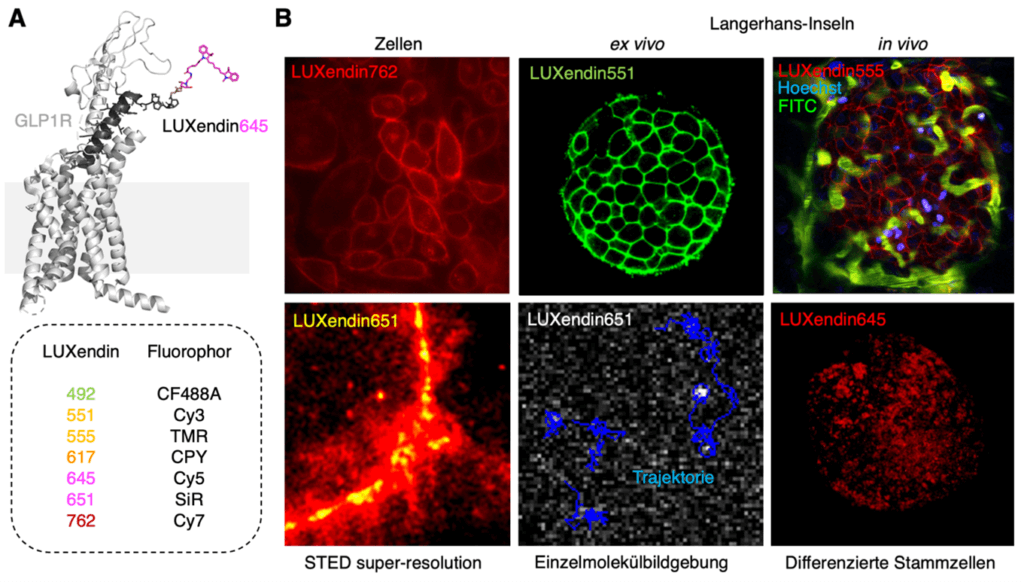
Abbildung 1: GLP1R Markierung mittels LUXendin. A) LUXendin645 besteht aus einem hoch-affinen, Fluorophor-verlinkten Peptid, das spezifisch an den glucagon-like peptide-1 receptor (GLP1R) bindet, und mit verschiedenen Farben ausgestattet werden kann. B) Verschiedene fluoreszenzmikroskopische Aufnahmen von GLP1R mittels LUXendinen, in Zellen, Langerhans-Inseln (ein Mini-Organ in der Bauchspeicheldrüse), für die hochauflösende STED Mikroskopie und zur Einzelmolekülbildgebung.
Referenzen
[1] E. Schrödinger, „Was ist Leben?“ Piper Taschenbuch, 1989.
[2] R. Dawkins, 2002, https://www.ted.com/talks/richard_dawkins_militant_atheism
[3] P. Kruse, 2008, Interview im Rahmen der SCOPE_08.
[4] Grimm et al., JACS Au 2021.
[5] Roßmann et al., bioRxiv 2020.
[6] Ast et al., Nat. Commun. 2020
[7] Ast et al., JACS Au 2022.






 4c media
4c media